Kurihara M., Shiraishi A., Satake H., and Kimura A.P.
A conserved noncoding sequence can function as a spermatocyte-specific enhancer and a bidirectional promoter for a ubiquitously expressed gene and a testis-specific long noncoding RNA
Journal of Molecular Biology 426: 3069-3093
PubMed HUSCAP 生 物科学科HPの記事
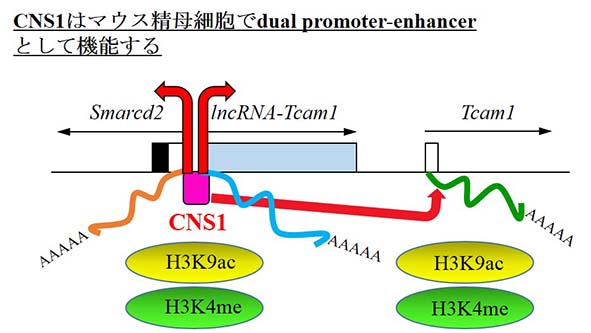
本研究はマウス精巣で特異的に発現する遺伝子の活性化メカニズムを明らかにする目的でスタートした。研究対象となった遺伝子はマウスの細胞接着タンパク質 をコードするTcam1(Testicular Cell Adhesion Molecule 1)遺伝子である。最初にこの遺伝子の発現パターンを確認するためノーザンブロット解析やin situ hybridization解析などを行った結果、Tcam1遺伝子が精母細胞特異的に転写されていることがわかった(Fig. 1)。次に、Tcam1遺伝子の調節配列の候補として進化的に保存された6つのCNS(Conserved Noncoding Sequence)を同定した。ヒストンH3K9アセチル化とヒストンH3K4メチル化を調べた結果、CNS1、CNS2、CNS3の3つ のゲノム配列がTcam1遺伝子を調節する可能性が考えられた(Fig. 2, Fig. 3)。そこで、マウス精母細胞由来のGC-2spd細胞を用いてこの3つのゲノム配列の転写調節活性を検証したところ、CNS1のみがGC-2spd細胞 特異的にTcam1プロモーターのエンハンサーとして機能することがわかった(Fig. 4, Fig. 5)。興味深いことに、この解析ではCNS1が強力な両方向性のプロモーター活性を持つこともわかり、実際に精巣においてクロマチン関連タンパク質をコー ドするSmarcd2遺伝子と新規のノンコーディングRNAであるlncRNA-Tcam1の転写を駆動するプロモーターになっていた(Fig. 5, Fig. 6)。CNS1が示すプロモーター/エンハンサー活性に重要な配列を明らかにするために、CNS1中で進化的に保存された配列に存在するSp1結合部位に 変異を入れた。その結果、Smarcd2遺伝子を駆動するプロモーター活性は低下したが、Tcam1遺伝子のエンハンサー活性とlncRNA-Tcam1 を駆動するプロモーター活性の低下は見られなかった(Fig. 7)。最後に、CNS1の示すプロモーター/エンハンサー活性がゲノムに組み込まれたDNA上でも見られるものなのか調べるために、Tcam1遺伝子の上 流6.9 kbの配列をEGFP(Enhanced Green Fluorescent Protein)遺伝子につないだコンストラクトを作成し、さらにその中からCNS1配列を削ったコンストラクトも作成した(Fig. 8a)。これらのコンストラクトをGC-2spd細胞のゲノム中に組み込んだ細胞クローンを作成して解析した結果、CNS1がクロマチン構造の中でもプロ モーター /エンハンサー活性を示すことがわかった(Fig. 8)。以上の結果は、マウスの精母細胞において、CNS1がTcam1遺伝子のエンハンサーとして機能できるだけでなく、Smarcd2遺伝子と lncRNA-Tcam1のプロモーターとしても機能するdual promoter-enhancerであることを示している(Fig. 9)。このようなdual promoter-enhancerと呼ばれる配列はこれまでニワトリでわずかに2例の報告があるだけで、本研究は哺乳類で初めてその存在を証明したもの で ある。