
はじめに
上野 宜久(農業生物資源研)
ある生命体(個体)が生命活動を営むためのほぼ全ての情報は、ゲノムDNAに書き込まれている。その情報とは、個体を構成する物質の情報や、これらをコントロールするための調節物質の情報である。ある場合には構成タンパク質が、ある場合には化合物を合成する酵素などのタンパク質が、またある場合にはこれらをコントロールするやはりタンパク質が、全てA, G, C, Tという4つの文字の並び(塩基配列)として書き込まれている。書き込まれた情報は読み出され、実行されて、生命活動は成り立つ。この情報の読み出しと実行を遺伝子発現と呼ぶ。
情報が読み出され実行されると言ったが、これは具体的にはどういうことだろうか?4つの文字に書き込まれている情報とは、もちろん全てではないが多くの場合タンパク質という具体的な物質の情報と考えてよい。タンパク質の種類は実に多様で、様々な化学反応を担う。タンパク質の情報はどんなタンパク質を作り出すかという情報とそのタンパク質をいつどこで作り出すか(働かせるか)という情報に大別される。20世紀後半から現在に至る分子生物学が解きほぐしてきた生命活動の実体とは、これら多様なタンパク質の働きとタンパク質を作り出す調節機構が大筋と考えてよいほど中心的なテーマであったし、今でもなお精力的に探求されている。ご存じのように、最近では技術革新の成果を取り入れ、ゲノム科学的アプローチが大規模になされた。どのような情報がゲノムに書き込まれているかを全て見てみようという近年のこのようなアプローチは、ある生物種の生命情報の全容を解き明かしたかのように勘違いされる初学者もひょっとするとみえるかもしれない。もちろん一生物種の全てのゲノムDNA情報を明らかにしたゲノム科学の貢献は計り知れないものであることを私たちは忘れたりはしていないが、文字列を明らかにしても、そこに書き込まれた暗号の意味を理解したとは言えないことを確認しておきたい。
先に、ゲノム情報の読み出しと実行を遺伝子発現と呼ぶと言ったが、もう少し踏み込んでみよう。具体的に遺伝子発現においては、多くの場合、タンパク質そのものをコードしている領域(翻訳領域)とその前後を含む比較的短い領域のゲノムDNAのA, G, C, Tの並びが、ほぼ完全にコピーされた並びのA, G, C, Uの4文字(塩基)が並んだRNAがその領域から合成される(転写)。このRNAの塩基配列は3塩基ずつの三つ組み暗号として意味をなし、それぞれに対応するアミノ酸が並んで連結され、タンパク質が合成される(翻訳)。(このDNAからタンパク質への流れはフランシス・クリックがセントラルドグマと呼んだ概念にあたる。その後様々な例外が発見され、あえて呼ばれることは稀になったが、生命原理の中心的な仕組みであることには変わりない。)ここまでの説明で登場したRNAをメッセンジャーRNAという。これから問題にしたいのは、このタンパク質合成にいたるゲノムの読み出し実行がいつどこで行われるか、すなわち調節の機構がどうなっているかである。
現在、主要と考えられかつ知見が蓄積しているのは、いつ(また多細胞生物の場合はどの細胞で)RNAを合成するか(転写の調節)である。転写されるそれぞれの遺伝子には固有の制御があるが、基本的な仕組みは共通の考え方で理解されている。転写を調節する情報(ゲノムDNAの配列)は、転写される領域の近傍にあることが多い。その配列情報に基づいて、転写を制御する複数のタンパク質が、複雑に相互作用しあってON/OFFを調節する。ここで簡単に紹介した基本的な転写調節の概念は、生物が普遍的に用いており、これで遺伝子発現は十分説明できるのではないかと錯覚すら覚えるほどシンプルで美しい。
では、実際に細胞の遺伝子発現は転写調節だけで成り立つかというと、現実はそれほど単純ではない。転写以外の調節系(総じて転写後制御(機構)とも呼ばれ、ここでは以後このように呼ぶ)が、極めて豊富に存在しており、かつ重要な役割を担っている。やっかいなこと(?)に、転写後制御は遺伝子ごとに極めて多様なのである。それ故、どの遺伝子にも適用できるような機構というのは現在のところ知られておらず、重要性についても様々な見解がある。タンパク質そのものの安定性等を制御する機構はとりわけ翻訳後制御と分類され、このような機構は別に考えるのが一般的なので、転写後制御はRNAの関わるシステムである。また、多様であるが故に研究対象も個別にならざるを得ない事情も生まれる。そんな中で、私たちは植物を材料に転写後制御に興味を持って研究を続けている。
RNAは、配列の書き換え(RNA編集)や、つなぎかえ(スプライシング)が起こる。生命進化の謎を解く鍵が潜んでいるかもしれないこれらの現象について、中村崇裕博士および大谷美沙都博士がそれぞれ興味深い応用研究等を交えてわかりやすく解説する。また、RNAは別のRNAによってその機能発現がコントロールされる標的でもある。近年爆発的に理解のすすんだものに小分子RNAがあり、これについて都筑正行氏・竹田篤史博士が解説する。これについては、相当数の総説/解説が世に出ているが、なかでも主役の一つであるマイクロRNAについて、わかりやすく簡潔でかつ理解の助けになる秀逸なマイクロRNA分類を採用・解説している。RNAは、タンパク質などの他の分子同様に作られる(転写)量と分解される量の差し引きでその総量が決定されるので、安定性に関する理解は極めて重要である。この重要なRNAの安定性について鈴木悠也氏・千葉由佳子博士が解説する。
植物は、食料という重要課題を負っているだけでなく、様々に魅力的な現象を持ち、基礎科学の面からも興味深い研究対象なのである。一例として細胞の分化の可塑性をあげたい。山中伸弥京都大学教授とJohn Bertrand Gurdonケンブリッジ大学教授の「成熟した細胞に対してリプログラミングにより多能性(分化万能性)を持たせられることの発見」によるノーベル医学生理学賞受賞の朗報は記憶に新しい。このような十分分化した体細胞が分化全能性(分化万能性)を獲得する(脱分化する)という現象が、植物では遺伝子導入という高度な技術を必要とせず容易に可能であることが半世紀以上も前から発見されていた。このことが語られる時にどのような視点であるかは語り手次第であるが、「ムリだろう!」と思われるような技術に対し「可能かもしれない」という希望を、研究対象としての植物はこれまで与え続けてきた。そのように、私たちはとらえている。
分子生物学とは、複雑な生命の現象の重要な(であろう)一側面を巧妙に切り出しシンプルに示す営みであると言う事もできる。そして、示すモデルが広く適用できる研究成果ほどそのインパクトは高く、特殊な現象が対象である場合には枝葉末節と扱われることもある。普遍性の示されていない転写後制御機構研究の少なくとも一部は、とても面白いにも関わらずそのような(普遍的でないというような)扱いを受けるであろうリスクを背負っているのも認めざるを得ない。しかし、掘り下げなければ掘り出せない。GFPの研究(下村修博士他2人、2008年ノーベル化学賞)がこれほどまでに分子生物学に貢献するものと、1960年代にどれほどの人が想像できたかということに想いを馳せると道半ばで希望を捨てる事は、やはりできないのである。
RNAにはここでは解説しきれない様々な性質が知られていることを申し添える。
RNA編集
中村 崇裕(九州大学)
RNA編集とは、転写後の遺伝子発現調節機構の一つであるが、他のRNAレベルでの制御と大きく異なる点は、DNAに記録された遺伝情報を「編集」、すなわち異なる遺伝情報に書き換えるところにある。
RNA編集はセントラルドグマを逸脱する生物現象であるが、1986年に発見されて以来、多くの真核生物で行われていることが明らかになってきた。ほ乳類のRNA編集異常は疾病研究との関係から多くの注目を集め、その分子機構が活発に研究されている。
RNA編集によってRNA中の特定の箇所の塩基が異なる塩基に置換、修飾、欠失、挿入、することで、DNAに記された遺伝情報と異なる蛋白質になったり、RNA編集によって遺伝子の発現レベルを変化する現象が見つかっている。多くの場合、RNA編集は、機能的な蛋白質を作るのに必要なRNA段階の制御として知られている。tRNA、rRNA、mRNA、miRNAなど、様々なRNA分子種がRNA編集を受けることが知られている。
以下にこれまでに発見されているRNA編集、その後にRNA編集の起源と進化、について紹介したい。
RNA編集の種類
挿入・欠失型のRNA編集 挿入・欠失型のRNA編集は原生動物のトリパノソーマで発見された最初のRNA編集である。このRNA編集では、編集前のRNA前駆体と一部相補的な配列を持つガイドRNAが2本鎖を形成し、複数の蛋白質が含まれる複合体(editosome)が結合することで、ガイドRNAの塩基配列を鋳型に用いて塩基の挿入または欠失を行う。塩基挿入により、フレームシフトが生じ、DNAにコードされる遺伝子と異なる蛋白質が生じる。
C-to-U置換のRNA編集 このRNA編集は、RNA中の特定のシチジン(C)をウリジン(U)に脱アミノ化することによって行われる。ヒトでは、肝臓で働くアポリポ蛋白質BはRNA編集を受けないが、腸で働くときにRNA編集を受ける。このRNA編集によって読み枠途中で停止コドンが生じ、最終的な遺伝子発現産物が器官によって異なる働きをすることが知られている。C-to-U置換のRNA編集は、植物のオルガネラ遺伝子によく見られる(後述の植物のRNA編集を参照)。
A-to-I置換のRNA編集 このRNA編集は、二本鎖RNA領域で起きる。Adenosine deaminaseがアデノシン(A)を脱アミノ化することで、イノシン(I)を生じる現象で、線虫からほ乳類まで幅広く保存されている。しかし、植物では相同な酵素がないとされている。RNA編集によってアミノ酸配列が置換する場合もあるが、ほとんどの標的塩基は非翻訳領域やnon-coding RNA中にあり、その生理学的な意義はほとんど分かっていない。最近、non-coding RNAの一つであるmiRNAの20%において、A-to-I置換のRNA編集が生じることが明らかにされ、このRNA編集の異常によってがん細胞における抗がん剤抵抗性や転移能が亢進されることが示唆されている。
植物のRNA編集 植物では、ミトコンドリアおよび葉緑体で起きるC-to-U、まれにU-to-C置換型のRNA編集しか見つかっていない。しかし、そのRNA編集箇所は非常に多く、例えば、維管束植物の葉緑体で約30箇所、ミトコンドリアで500箇所以上のRNA編集が見つかっている。典型的なケースとして、ツノゴケ葉緑体の76遺伝子中に約1000箇所のRNA編集部位が見つかっている。多くの場合、これらRNA編集部位は蛋白質コード領域に含まれており、編集後に機能的な蛋白質遺伝子となる。機能多様化というよりも、ゲノム上の変異を修正するような方向でRNA編集が起きる場合がほとんどである。
植物オルガネラのRNA編集のメカニズムは未だ解明されていないが、編集部位の5’上流配列を配列特異的なRNA結合蛋白質であるPPR蛋白質が認識し、触媒酵素を呼び込む、と考えられている。触媒酵素の分子実体はいまだわかっていない。PPR蛋白質遺伝子は植物のみで非常に大きなファミリーを形成しており、オルガネラ遺伝子発現に遺伝子特異的に作用することが明らかになりつつある。
ウイルスのRNA編集 一部のウイルス(パラインフレエンザ、麻疹、ムンプス(おたふく風邪)、など)では、RNA編集により、多様な蛋白質が生じたり、転写物の安定性が向上したりする。ウイルスのRNAは、ウイルスゲノムがコードするRNA依存型RNA転写酵素によって転写されるが、伸長途中に特定の箇所でいったん停止する。停止したときに1個もしくは2個のG(またはA)を取り込むことで、異なる蛋白質をコードするRNAを生成する。また、RNAの3’末端に鋳型非依存的に数百塩基のAが付加され、転写物が安定化する。
RNA編集の進化と起源
C-to-U、もしくはA-to-I型のRNA編集を触媒する酵素は、塩基そのものを生成する代謝経路で働く酵素とよく似ている。原核生物にも該当する酵素があるが、これはRNA上の塩基置換に働くことは出来ない。そのため、真核生物の進化の過程で、RNA編集酵素として働くように新しい機能を獲得したと考えられる。また、植物と動物では、RNA編集に関わる蛋白質は異なり、それぞれが独立して獲得したと考えられる。トリパノゾーマの挿入・欠失型のRNA編集は全く異なるプロセスで行われる。
このようにRNA編集は、それぞれの生物で独自に獲得、進化してきたと考えられる。また、真核生物のみで確認されており、原核生物では行われないことも興味深い。植物では、水性の光合成生物ではRNA編集は見つかっておらず、陸上へと生活環境を移した陸上植物のみでRNA編集が観察されるため、太陽光暴露によるDNA損傷を修復するシステムとしてRNA編集が発達してきたとする説が有力である。
RNA編集は、DNAが生命の遺伝情報を司る、というセントラルドグマに反するRNA修飾である。その意義やメカニズムをさらに研究することで、新たな生物への理解が得られると考える。
カスタムRNA結合蛋白質の開発
近年、様々な解析より明らかになった核酸結合性の蛋白質因子を用いて、意図する配列に結合する技術が確立、利用されている。この配列特異的な結合を利用することで、標的とする核酸(DNAまたはRNA)の細胞内局在の解析、標的とするDNA配列の除去、またはその下流に存在する蛋白質コード遺伝子の発現の制御(活性化、または不活化)、が可能になりつつある。核酸性素材を用いると多くの場合、標的分子の不活化にしか用いることができず、特にmiRNAの場合、RNA成分であるがゆえの不安定性、RNA干渉システムを有する器官のみで利用可能、などいくつかのデメリットがある。一方、蛋白質成分を用いた場合、核酸認識部分に様々な作用ドメインを融合することで、標的とする核酸分子に様々な効果を与えることができる。
DNAに作用する蛋白質性因子として、ジンクフィンガー蛋白質やTAL effector、が蛋白質工学的材料として研究および開発が行われており、ゲノム改変による新たなモデル細胞の樹立、遺伝病の治療、有用作物の作出、などに適応されており、ゲノム編集という新しい分野が確立した。
しかし、RNAに特異的に作用する蛋白質性因子の開発はいまだ非常に限定されている。これは、一般的に蛋白質を構成するアミノ酸配列が有するRNAとの親和性および結合RNA配列との法則性がほとんど明らかになっていない、もしくは法則性が見いだせない、ためである。例外的に、pufモチーフ(38アミノ酸)の複数個の繰り返しで構成されているプミリオ蛋白質は、pufモチーフ1個がRNA1塩基に結合することが示されている。pumilio蛋白質を用いた新規RNA結合特性をもつ蛋白質、およびRNA結合特性の改変技術が試みられている。しかし、puf モチーフはそのモチーフが非常に保存され、かつ存在数が少ない。また、1アミノ酸置換により、蛋白質の活性そのものが損なわれることが多い。そのため、限られたRNA配列に作用する蛋白質因子の創成にしか用いられていない。様々な配列に様々な親和性で結合するRNA結合因子の自由度の高い設計には、新規のRNA結合モチーフの発見、利用が必要である。
九州大学の中村は、植物のみで500個もの大きなファミリーを形成する新規蛋白質、pentatricopeptide repeat (PPR)モチーフを有するタンパク質、に着目し、カスタムRNA結合蛋白質の開発を進めている。
PPR蛋白質は核コードであるが、専らオルガネラ(葉緑体とミトコンドリア)のRNAレベルでの制御(切断、翻訳、スプライシング、RNA編集、RNA安定性)に遺伝子特異的に作用する。PPR蛋白質は保存性の低い35アミノ酸から成るPPRモチーフが平均10個連続したモチーフ構造を有している。最近、PPRモチーフとRNAが1対1の対応関係で結合すること、PPRモチーフを構成する35アミノ酸のうち、特定の3カ所のアミノ酸が結合する塩基の種類を決める「RNA認識コード」を司ることを明らかにした。このRNA認識コードの発見により、植物の内在性PPR蛋白質の標的RNA予測、カスタムRNA結合蛋白質の開発に資する基礎技術基盤が確立された。現在、PPRを素材としたカスタムRNA結合蛋白質の開発を進めており、学術分野、医療分野、農業分野を含む全ての生物系産業、またナノデバイスとして工学系産業への利用が期待できる。
メッセンジャーRNAの旅の始まり〜転写から核外輸送まで〜
大谷 美沙都(奈良先端大学院大学)
生物の遺伝情報は、ゲノムDNAに遺伝子配列として書き込まれている。これらの情報はRNAポリメラーゼと呼ばれる酵素によってRNAへと転写された後、機能性RNAとして、あるいはタンパク質へと翻訳されて、さまざまな生体機能を果たしていく。本項では、とくにタンパク質翻訳の鋳型となるメッセンジャーRNA(messenger RNA; mRNA)に焦点を当て、mRNAが受ける修飾について概観する。
ゲノムDNAからの転写と構造修飾
タンパク質をコードする遺伝子は、一般的に、転写制御に関わる上流転写調節域、転写開始シグナル配列、タンパク質翻訳領域、転写終結シグナル、などの機能的構造からなっている(図1)。
転写の頻度や開始箇所は遺伝子そのものが持つ転写調節配列(シス配列)やその他の転写制御因子(トランス因子)によって制御されており、最終的に遺伝子上に呼び込まれたRNAポリメラーゼII複合体が活性化されることによって、mRNAの合成が始まる(図2; Taatjes et al. 2004 Nature Rev. Mol. Cell Biol. 5: 403-410)。
ゲノムDNAから転写されたmRNAは、直後には前駆体と呼ばれる状態である(図3)。これは、mRNAは転写されてから最終的にタンパク質鋳型となるまでの間に、さまざまな構造的・化学的修飾(一連の過程をまとめてプロセシングとよぶ)を受ける必要があるためで、各修飾ステップがそれぞれ遺伝子発現調節段階となっていることが知られている。
転写直後に最初にmRNA前駆体が受けるのが、末端構造修飾である。植物を含む真核細胞の場合、mRNAの5’末端(mRNAはゲノムDNAのアンチセンス鎖を鋳型として5’→3’方向に転写される)には5’-メチルグアノシンが付加され、特有のキャップ構造が形成される(図3; Ghosh and Lima 2010 Wiley Interdiscip. Rev. RNA 1: 152-172)。さらに転写終結後のmRNAの3’末端にはポリA付加酵素によって連続したA(アデニル酸)が付加され、ポリAテイルを形成する(図3; Colgan and Manley 1997 Genes Dev. 11: 2755-2766)。キャップ構造にはキャップ結合複合体と呼ばれるタンパク質複合体が、ポリAテイルにはポリA結合タンパク質が結合し、どちらもmRNAの安定性やプロセシング進行、核外輸送、さらには翻訳開始調節に重要であることが分かっている。 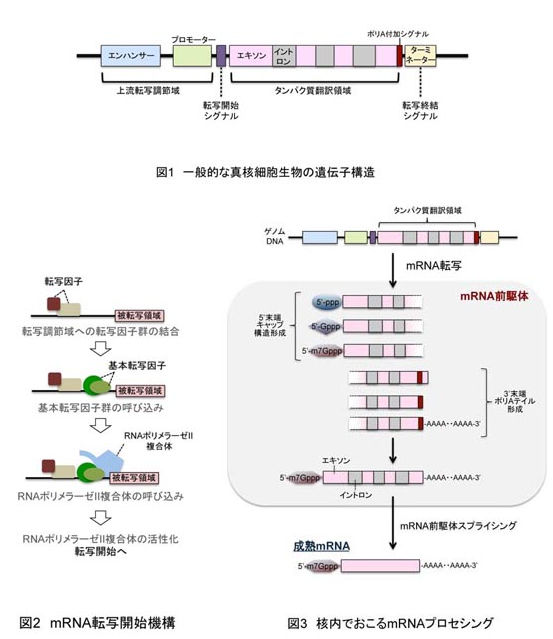
mRNA前駆体スプライシング
真核生物の場合、多くの遺伝子はタンパク質コード領域内にエキソンとイントロンと呼ばれる2種類の領域を含んでいる。エキソンはアミノ酸をコードする配列領域であり、イントロンは非コード領域である。つまり機能的なタンパク質を生成するためには、イントロン領域を除去した翻訳鋳型を作り出す必要がある。これがmRNA前駆体スプライシング(pre-mRNAスプライシングともよばれる)である(Wahl et al. 2009 Cell 136: 701–718)。
mRNA前駆体スプライシングは、スプライソソームと呼ばれる巨大なRNA-タンパク質複合体によって行われる。スプライソソームは5つの中心的な核内低分子リボ核酸タンパク質(snRNAと呼ばれる核内低分子RNAと特異的に結合するタンパク質からなり、snRNPとよばれる)と、多数の関連タンパク質からなっており、スプライシング進行にともなって構成因子が複雑に入れ替わりながら機能する。スプライシングの基本機構は酵母を使った研究によって明らかにされてきた。酵母研究を元にしたモデルによると(図4)、スプライシング反応はまずU1 snRNPによるイントロンの5’末端認識から始まる。続いてイントロン内に存在するブランチ部位と呼ばれる配列にU2 snRNPが結合し(A複合体)、さらに他の3種のsnRNPがU4/U6-U5 tri-snRNPとして会合した状態で呼び込まれ(B複合体)、U1 snRNPとU4 snRNPの解離を経た後、活性型スプライソソームを形成する。活性型スプライソソームは2段階のトランスエステル反応を触媒して、エキソン同士の結合と、ラリアット構造化したイントロンを作り出す。結合したエキソンはmRNAとして直ちにスプライソソームから解離し、さらにラリアット構造化したイントロンはスプライシングの最終段階でスプライソソームから解離する(図4)。
こうした一連の反応は、U snRNP構成因子と50以上もの非snRNPタンパク質群のダイナミックな構成的・構造的変化と連動しながら行われ、動物や植物といった多細胞生物では、酵母に比べてさらに多種タンパク質がスプライシング反応に関わっていると考えられている(Will and Lührmann 2011 Cold Spring Harb. Perspect. Biol. 3: a003707)。また、スプライシングに関わる因子の一部は反応終結後もmRNAと結合し続け、核外輸送や翻訳開始調節などに引き続き機能することも分かっている。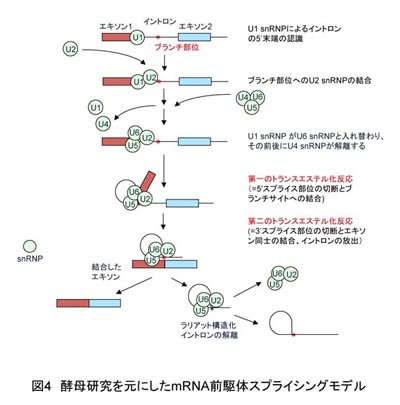
スプライシングがもたらすもの
mRNA前駆体スプライシングは、上述のように遺伝子内からイントロンを切り出し、タンパク質合成のための翻訳鋳型を作り出す仕組みである。この機構は遺伝子発現の調節段階となりうると同時に、とくに真核生物にとって、それ以上の重要な役割をもっていることが、明らかとなってきた。
2001年、ヒトゲノムの配列解析結果が発表されその後解析が進むにつれ、mRNA種数から予測されていた数(約10万種〜)よりも、実際に見つかった遺伝子総数(2万5千個前後)が少なかった点に注目が集まった(IHGSC 2001 Nature 409: 934−941; IHGSC 2004 Nature 431: 931-945)。これはつまり、1つの遺伝子から複数種のmRNAが作られていることを意味しており、実際にヒトの場合、90%以上の遺伝子が複数のmRNAを作り出しているとも言われている。こうした状況を生み出しているのが、選択的スプライシングとして知られてきた仕組みであった。
選択的スプライシングとは、mRNA前駆体が異なるパターンのスプライシングを受け、1つの遺伝子から複数種のmRNAが作られることである(図5; McManus and Graveley 2011 Curr. Opin, Genet. Dev. 21: 373–379)。前項2)で述べたように、スプライシングの際にはmRNA前駆体に存在するイントロン5’末端やブランチ部位の配列が部位認識に重要な役割を果たしているが、酵母ではこれらの認識配列がほぼ100%保存されているのに対して、動物や植物では保存性が低くなっている。このことが、スプライシング部位選択にバリエーションを持たせることになり、選択的スプライシングが可能になっている。さらにはこうした認識配列選択に影響を与えるような配列(エンハンサー配列やサイレンサー配列)がエキソン・イントロン内に存在し、積極的な選択的スプライシング制御が行われていることも知られている(Wang and Burge 2008 RNA 14: 802-813)。
選択的スプライシングの結果生まれるmRNAのバリエーションとしては、活性ドメインや局在シグナルの付加・欠如によってタンパク質機能性の変化をもたらす場合や、mRNA自体の安定性に関わる領域の付加・欠如を制御して発現量自体を変化させる場合などがある。遺伝子によっては、こうした選択的スプライシングによって、時と場合によった複数の遺伝子機能を果たしているものもある。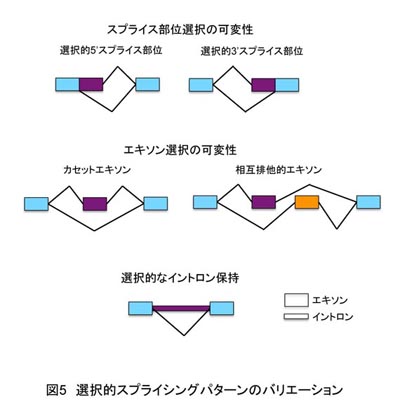
そして核外へ; 新たな旅の始まり
5’末端へのキャッピング、mRNA前駆体スプライシング、3’末端へのポリAテイル付加といった一連の修飾を受けてプロセシングが終了した成熟mRNAは、核膜孔を通過して核外へと輸送され細胞質に移動し、タンパク質翻訳の鋳型となる。これまでに各々のプロセシング機構と核外輸送とに共通して関与しているタンパク質が多数知られており、実際にはこれら各プロセシング・核外輸送機構は複雑に共役しながら進行していると考えられている。この共役を通して、真核細胞は正常なプロセシングを受けた成熟mRNAの核外輸送の効率化と同時に、異常mRNAや未成熟なmRNAを核内に留めるRNA品質管理を実現しているのであろう。
成熟mRNAは複数タンパク質と結合したmRNP(mRNA-タンパク質の複合体)として核外へと輸送される。具体的には、核膜に存在する核膜孔複合体と呼ばれる巨大な孔構造体とmRNPに含まれるタンパク質が相互作用して孔が押し広げられ、mRNPの通過が可能になる(Rodriguez et al. 2004 Biol. Cell 96: 639–655)。通過後、mRNPを構成していたタンパク質の一部はmRNPから解離、また一部はそのまま残留し、翻訳開始に向けて、mRNAは細胞質でのさまざまな調節制御を受けることになる。
細胞質でのさまざまな翻訳制御機構、またmRNA分解機構については別項に詳細を譲るが、細胞質に輸送された成熟mRNAはやみくもに翻訳鋳型となるのではなく、複雑かつ積極的な調節・制御を受けて最終産物であるタンパク質に翻訳され、最終的に分解されることが分かってきている。すなわち、mRNAは誕生(転写)直後からその死(分解)まで、絶えず種々の因子に囲まれ、相互作用を繰り返し、修飾を受け、輸送され、きわめて忙しい一生を過ごすと言える。
なお、本項で概説した内容は、主に酵母やヒトの研究成果によるものである。mRNAの転写〜核外輸送までの機構については、基本的に真核生物間ではほぼ保存され、類似の分子装置が働いていると信じられている(Simpson et al. 1991 Nucleic Acids Res. 19: 5213–5217; Wang and Brendel, 2004 Genome Biol. 5: R102; http://www.plantgdb.org/SRGD/index.php)が、実は植物細胞において実験的に詳細が検討された事例は少ないのが現状である。これは、植物細胞での適当なin vitro実験系の開発の遅れや、染色体倍加にともなう関連遺伝子数の増大など、植物研究ならではの困難さを反映したものと言えよう。しかし同時に、植物特有の仕組みの存在を示唆する研究事例も存在している。今後の当該領域における植物研究の発展に、大いに期待したいところである。
植物のmicroRNA
竹田 篤史(立命館大学),都筑 正行(東京大学)
small RNAとは?
small RNAとは、20〜30塩基程度の短いRNAであり、真核生物で遺伝子発現抑制に関与している。small RNAが関与する遺伝子発現抑制は、配列特異的である。small RNAとの相補性に依存して標的遺伝子が決定される。small RNAはArgonaute(AGO)と呼ばれるタンパク質に取り込まれる。AGO-small RNA複合体は、mRNA切断、翻訳抑制、ゲノムDNAのメチル化誘導などによって配列特異的な遺伝子発現抑制を行なう。植物におけるsmall RNAの生物学的意義は、分化・発生の制御、環境応答、ウイルス抵抗性、トランスポゾンの転移・増殖の抑制など非常に多岐に渡る。以下にモデル植物アラビドプシスにおけるsmall RNA経路の分子機構とその役割について概説する。
small RNAの種類とその生合成
small RNAは、生合成過程の違いから、microRNA(miRNA)、small-interfering RNA(siRNA)、PIWI-interacting RNA (piRNA)に大きく分類される。このうち、piRNAは植物には存在しない。
・miRNA
miRNAは内在性のsmall RNAであり、相補的な配列を持つmRNAに作用することで、標的遺伝子の発現を負に調節する。植物のmiRNAの長さは約20〜22塩基長である。miRNAにはmiR156、miR172のように通し番号が付けられている。全生物種のmiRNAのデータベースとしてmiRBase(http://www.mirbase.org/)、アラビドプシスのmiRNAのデータベースとしてAT Small RNA(http://mpss.udel.edu/at_sbs/index.php?menu=../common/web/GeneQuery.php?SITE=at_sbs)が挙げられる。
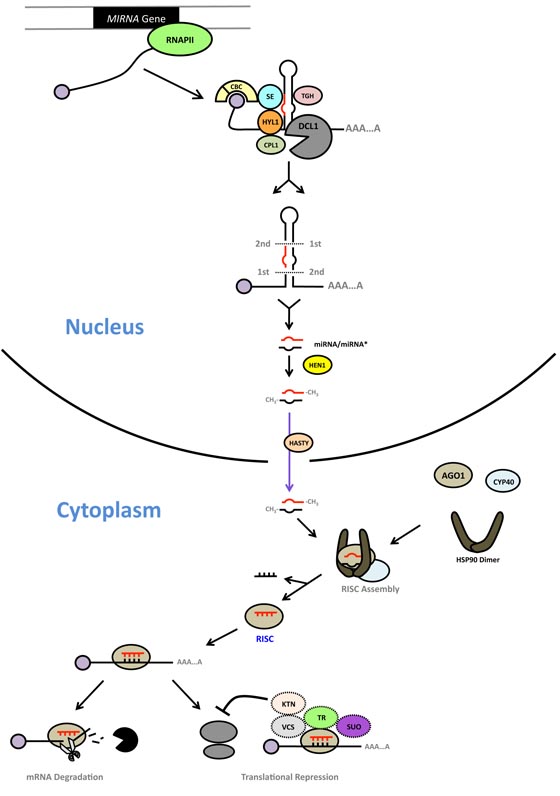 miRNAの生合成過程を図1に示した。miRNAの前駆体(Pri-miRNAと呼ばれる)は、mRNAの場合と同様にRNAポリメラーゼIIによって転写される。Pri-miRNAは、逆位反復配列を持つため、転写されるとヘアピン構造を取る。このヘアピン構造からmiRNAを切り出す主要タンパク質が、Dicer-like 1(DCL1)である。DCL1はHYPONASTIC LEAVES 1 (HYL1)、SERRATE (SE)タンパク質などと共同して働き、まずPri-miRNAからヘアピン構造(Pre-miRNAと呼ぶ)を切り出し、次にPre-miRNAから二本鎖miRNAを切り出す。このうち、AGOに取り込まれて働く鎖をガイド鎖、もう一方の鎖をパッセンジャー鎖と呼ぶ。パッセンジャー鎖はmiRNA*と表記する。miR172を例にすると、ガイド鎖をmiR172、パッセンジャー鎖をmiR172*、二本鎖miRNAをmiR172/miR172*と表記する。DCL1、HYL1、SEは核内のDicing-bodyと呼ばれる場所に局在している。
miRNAの生合成過程を図1に示した。miRNAの前駆体(Pri-miRNAと呼ばれる)は、mRNAの場合と同様にRNAポリメラーゼIIによって転写される。Pri-miRNAは、逆位反復配列を持つため、転写されるとヘアピン構造を取る。このヘアピン構造からmiRNAを切り出す主要タンパク質が、Dicer-like 1(DCL1)である。DCL1はHYPONASTIC LEAVES 1 (HYL1)、SERRATE (SE)タンパク質などと共同して働き、まずPri-miRNAからヘアピン構造(Pre-miRNAと呼ぶ)を切り出し、次にPre-miRNAから二本鎖miRNAを切り出す。このうち、AGOに取り込まれて働く鎖をガイド鎖、もう一方の鎖をパッセンジャー鎖と呼ぶ。パッセンジャー鎖はmiRNA*と表記する。miR172を例にすると、ガイド鎖をmiR172、パッセンジャー鎖をmiR172*、二本鎖miRNAをmiR172/miR172*と表記する。DCL1、HYL1、SEは核内のDicing-bodyと呼ばれる場所に局在している。
アラビドプシスにはAGO1〜AGO10の10種類のAGOホモログがコードされている。AGOタンパク質とsmall RNAを含み、遺伝子発現抑制を行う複合体をRNA-induced Silencing Complex (RISC)と呼ぶ。AGO1を含むRISCは、AGO1-RISCと表記する場合もある。AGO1-RISC形成にはCyclophilin 40 (CYP40)とHSP90が重要な役割を果たしている。多くのmiRNAはAGO1に取り込まれて標的mRNAの切断に働く。ago1のnull変異体はseedling致死の表現型を示す。つまり、AGO1-RISCによる遺伝子発現制御が植物の発生・生長に必須なことが明らかである。AGO1中には5'末端がUのsmall RNAが多く取り込まれている。そのためか、多くのmiRNAの5'末端はUである。実験的に、AGO2、AGO5、AGO7、AGO10の中にmiRNAが取り込まれることが示されている。このうち、AGO7はmiR390を、AGO10はmiR165とmiR166を特異的に取り込む。また、AGO2は5'末端がAのAGO5は5'末端がCのsmall RNAを好んで取り込む。
一般的に、植物で保存されているmiRNAに対しては、非常に相補性が高い標的mRNAが存在している場合が多い。これらの標的mRNA上では主にmRNAの切断によって遺伝子発現抑制を行っていると考えられる。ちなみに、miRNAと相補的な部位での内在性標的mRNAの切断は、植物で最初に発見された現象である。
miRNA種類とその標的遺伝子
アラビドプシスでは、miRNAに対して相補性が高いmRNAが存在していたため、動物と比較して早くから標的mRNAが予測されてきた(しかし、植物でも相補性の低いmRNA上でmiRNAが重要な作用をしている可能性は残っている。)。miRNAの標的遺伝子には、転写因子、miRNA経路で働くDCL1・AGO1・AGO2などの遺伝子、TAS non-coding RNAなどがある。表1に主なmiRNAとその標的遺伝子をまとめた。以下に主なmiRNAとその標的遺伝子について概説する。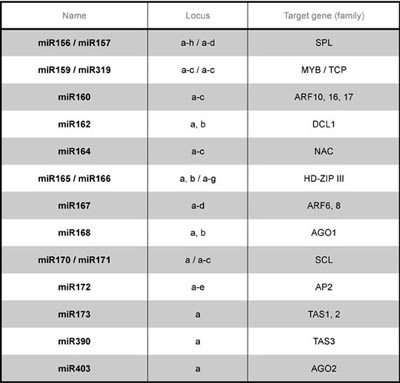
・miR156, miR157, miR172
miR156/157とmiR172は、経時的な発現変化により植物体の発生時期を調節していることが知られている。miR156/157は、発生初期に高い発現量を示し、発生が進むに連れて発現量が下がっていく一方、miR172は発生初期には発現量が低く、栄養成長期から成熟期、花成期へと進むに連れて発現量が上がる。miR156の過剰発現体は花成期への移行が遅くなり、miR172の過剰発現体は逆に早くなること、また逆にmiRNAの標的部位を改変した植物体や、miRNAの機能を阻害するmimicコンストラクト(Todesco et al. 2010 PLoS genetics 6: e1001031)を形質転換した植物体は反対の表現型を示すことから、これらのmiRNAは植物が正常に発生段階を移行するのに重要な働きを持つと言える。加えて、miR156のターゲットの一つであるSPL9がMIR172b遺伝子のプロモーター領域に結合することで発現を促していることがわかっており(Wu et al. 2009 Cell 138: 750-759)、miR156の減少に伴ってmiR172の発現が上昇することになることから、この2種のmiRNAは密接な関係を持っていると言える。
・miR159, miR319
miR159とmiR319は配列が似ており、標的遺伝子も一部共有しているが、主にmiR159がMYBファミリーを、miR319がTCPファミリーを抑制しており、役割を分担している(Palatnik et al. 2007 Dev. Cell 13: 115-125)。miR159ab変異体は葉の歪曲という目に見える明らかな表現型を示し(Allen et al. 2007 PNAS 104: 16731-16736)、miR319の過剰発現体も葉の歪曲を示すことから、葉の正常な発生に重要であることがわかっている。
・miR160
標的遺伝子はARF10、ARF16、ARF17で、初期段階からその後まで植物体の幅広い発生段階で重要な役割を持つ。ARF10、ARF16、ARF17のmiRNA結合部位改変体は、葉がノコギリ状になる、アブシジン酸に過剰に応答し発芽率が低下する、根冠の形成に異常が起こる、花序の形成が異常になる、などの表現型を示す(Liu et al. 2007 Plant J 52: 133-146、Wang et al. 2005 Plant Cell 17: 2204-2216、Mallory et al. 2005 Plant Cell 17: 1360-1375)。また、オーキシンが、MIR160a遺伝子の3’側に存在する調節領域を通してmiR160の発現を上昇させることもわかっている(Liu et al. 2010 Plant J 62: 416-428)。
・miR164
miR164はCUC1、CUC2などのNAC-Like転写因子ファミリーを抑制することで地上部の発生に関わっている。3つある遺伝子座のうちmir164c変異体は弱い発生異常を示す一方、mir164abcの三重変異体は雌蘂や雄蘂の個数が増えるなど重い表現型を示し、miR164の3つの遺伝子座はお互い冗長的に働いていることがわかっている(Sieber et al. 2007 Development 134: 1051-1060)。また、茎頂分裂組織の維持に必要なSTMがCUC1、CUC2、MIR164aの発現を活性化させることもわかっている(Spinelli et al. 2011 Plant Phys. 156: 1894-1904)。
・miR167
miR167の標的遺伝子であるARF6、ARF8はmiR160の標的遺伝子と同じファミリーでありながら、異なった役割を持ち、植物の生殖系列で強い発現が見られる。ARF6、ARF8それぞれの遺伝子にmiR167による発現抑制を受けない変異を入れた植物体は、どちらの場合も不稔であり、miR167は胚珠と葯の発生に重要な役割を果たしていることがわかっている(Wu et al. 2006 Development 133: 4211-4218)。
・miR165, miR166
この2種類は21塩基中1塩基のみ異なるため、同じファミリーとして扱われている。標的であるPHB、PHV、REVなどの複数のHD-ZIP III遺伝子を抑制することで、植物体の茎側、根側両方向の発生・分化を制御している。PHBとPHVの標的部位に突然変異が入った変異体は、地上部の背軸側が向軸側に変化してしまうという表現型を持つことからmiR165/166による発現抑制機構が地上部の器官形成に重要な役割を担うことを示している(McConnell et al. 2001 Nature 411: 709-713)。また、MIR165a、MIR166b 遺伝子はSHR、SCRなどの根における主要因子の下流に存在し、根の器官形成に関わることもわかっている(Carlsbecker et al. 2010 Nature 465: 316-321)。
・miR170, miR171
動植物を通じてmiRNAによる標的mRNAの切断が最初に観察されたのは、miR171によるSCL mRNAの切断である(Llave et al. 2002 Science 297: 2053-2056)。MIR171aの過剰発現体は上葉が減少し、MIR171cの過剰発現体は枝分かれの減少という表現型を示す(Fedoroff et al. 2010 Curr. Biol. 20: 37-41、Wang et al. 2010 Mol. Plant 3: 794-806)。また、mimicコンストラクトの形質転換体は、稔性の低下という表現型を示す。
・miR162, miR168, miR403
これらのmiRNAは、small RNAの遺伝子発現制御に直接関わるタンパク質の発現抑制を担っている。miR162はDCL1、miR168はAGO1 、miR403はAGO2をそれぞれ標的にしている。miR168の標的部位に変異を入れたAGO1遺伝子を形質転換した植物ではAGO1の量が増加し、植物は歪小化するという重篤な表現型を示す(Vaucheret et al. 2004 Genes Dev. 18: 1187-1197)。このことから、これらのmiRNAによって、small RNA経路の主要因子の発現量が適正に保たれることは、非常に重要であろうと考えられる。
・miR173, miR390
この2つのmiRNAは、植物に特有のTAS non-coding RNAを標的としている。TAS non-coding RNAは、ta-siRNA(後述)の生合成に必須な内在性のRNAであり、miR173、miR390によるTAS non-coding RNAの切断がta-siRNA生合成経路の起点となっている。
人工miRNAを利用した遺伝子発現抑制
応用として、人工miRNAを利用して目的の遺伝子を特異的にノックダウンする手法が一般的になってきた。アラビドプシスではMIR319aの骨格が広く用いられている。人工miRNAの設計は、WMD3 website(http://wmd3.weigelworld.org/cgi-bin/webapp.cgi)を利用して行うことが可能である。mRNA上の人工miRNA標的部位が高次構造を取っていたり、別のRNA結合タンパク質に保護されていたりする場合、人工miRNAはうまく働かない。そのため、複数種類の人工miRNAを用いて遺伝子発現抑制を試みるべきであろう。また、組織特異的プロモーターを利用して人工miRNAによる遺伝子発現抑制をする場合、miRNAの細胞間移行に注意する必要がある。
mRNA分解による遺伝子発現制御
千葉 由佳子(北海道大学),鈴木 悠也(北海道大学)
mRNA分解を介した遺伝子発現制御機構
生物は細胞内におけるmRNA量を厳密に制御することによって,変動する外部環境への適応を行い,生体内の恒常性を維持している。このような遺伝子発現調節の解析は,しばしばクロマチンの構造解析や転写因子の同定と言った,転写段階における発現調節に焦点が当てられがちであった。しかしながら実際のmRNA量の調節は転写と分解のバランスによって制御されており,ここ数年では小分子RNAの発見も伴い,mRNA分解を介した遺伝子発現制御の重要性が次々と明らかになっている。
植物における普遍的なmRNA分解経路
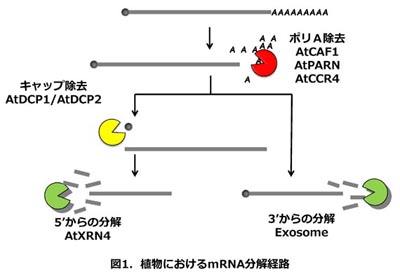
mRNAは5’末端にキャップ構造,3’末端にポリA鎖を持ち,分解を受けていないmRNAは,Poly(A)-binding protein (PABP) がポリA鎖に結合し,さらにPABPがキャップ結合因子と相互作用することによって,環状構造をとっている。この構造によって,mRNAはエキソリボヌクレアーゼによる分解から保護されていると考えられる。mRNA分解の多くは脱アデニル化酵素によるポリA鎖の除去に始まり,その後速やかに脱キャップ酵素により5’末端のキャップ構造が取り除かれて5’末端側から分解されるか,あるいはポリA鎖が除去された後に3’末端側からの分解を受ける(図1)。このようなmRNA分解経路に関する研究は酵母で最も進んでおり,これまでにそれぞれの段階に働く酵素が同定されている。これらの酵素は植物においても保存され,植物も似たような分解経路を持つと考えられている。ただし,植物に存在するPoly(A)-specific ribonuclease(PARN)というポリA除去酵素は酵母には存在せず,酵母とは異なる一面ももつ。
【1】植物における脱アデニル化酵素
酵母ではポリA鎖の除去を行う脱アデニル化酵素としてCarbon Catabolite Repressor 4 (Ccr4) / Ccr4 associated factor 1(Caf1)複合体や,Pab1p-stimulated poly(A) ribonuclease 2 / 3 (Pan2 / 3)複合体などが知られており,これらの欠失変異株では長いポリA鎖をもつmRNAが蓄積することが明らかになっている。なかでも,Ccr4 / Caf1を主要構成因子とする複合体が主要な脱アデニル化酵素として報告されており,変異株を用いた解析からCcr4がその複合体において中心的な役割を担っていることが明らかになっている。一方,多細胞生物においても複数の脱アデニル化酵素を持つことが明らかになっている(Dupressoir et al., BMC Genomics, 2, 9, 2001)。代表的なものとしてExonuclease-endonuclease-phosphatase(EEP)ファミリーに属するCcr4,NocturninおよびAngelと,DEDD-type nucleaseファミリーに属するCaf1, Pan2,およびPARNなどが報告されている。なかでもCcr4,Caf1,Pan2およびAngelは多くの真核生物で相同遺伝子が存在する。近年の研究から,一部の脱アデニル化酵素では発現の器官特異性が異なることなどが明らかになっており,脱アデニル化酵素の種類に応じて様々な役割を担っていることが推測されている。例えばアフリカツメガエルのNocturnin遺伝子は光周期依存的に発現が制御されており,また,そのタンパク質は桿体細胞や錐体細胞に強く局在することが明らかになっている(Baggs et al., Curr. Biol., 13, 189-198, 2003) 。このように真核生物は生体内に様々な種類の脱アデニル化酵素を持ち,さらにこれらをその目的に応じて使い分けることで,ある特定の成長段階あるいは環境ストレスに応じて標的となる遺伝子の発現を制御していることが考えられる。
モデル植物であるシロイヌナズナも,AtCaf1, AtPARN およびAtCCR4などの複数の脱アデニル化酵素を持つ。また,Caf1は11個の遺伝子から成るファミリー(AtCAF1a-k)を形成していることが明らかになっており,そのうちAtCAF1aおよびAtCAF1bは植物ホルモンの1つであるJasmonic acid(JA)によって発現が上昇する遺伝子として同定された(Liang et al., Cell Res., 19, 307-316, 2009) 。またAtCAF1aおよびAtCAF1bは酵母caf1突然変異株において酵母Caf1の機能を一部相補し,in vitroの実験系においても脱アデニル化酵素活性を有していることが明らかになっている。一方で標的遺伝子の単離を目的としたマイクロアレイを用いた解析により,AtCAF1aとAtCAF1bはアミノ酸配列において77%の相同性を示すにも関わらず,それぞれが特異的な標的遺伝子を持つことが示された(Walley et al., Plant Physiol., 152, 866-875, 2010) 。
AtPARNもまた脱アデニル化酵素活性を持つことが報告されている。突然変異株を用いた解析によってatparn突然変異株は胚発生致死になることが明らかになり,このことからAtPARNは胚発生に関わる遺伝子の発現制御を行っていることが示唆された(Chiba et al., Gene, 328, 95-102, 2004; Reverdatto et al., RNA, 10, 1200-1214, 2004) 。さらに,AtPARNの漏出突然変異株を用いた解析により,この変異株では内生のABA量が増加し,そのために外生のABAに対して高感受性となることが明らかになった(Nishimura et al., Plant J., 44, 972-984, 2005) 。このように植物の脱アデニル化酵素は,植物ホルモンに対する応答や胚発生など,植物特有の現象に関わる遺伝子の発現制御にも深く関わっていることが示されている。
【2】植物における脱キャップ酵素と5’-3’エキソリボヌクレアーゼ
脱アデニル化されたmRNAは脱キャップ酵素により5’末端のキャップ構造が外され,引き続き5’-3’エキソリボヌクレアーゼによって5’側からの分解を受ける。この段階に働く酵素として,シロイヌナズナではAtDCP1 / AtDCP2およびVaricose(VCS)から成る複合体が脱キャップ酵素として報告されている。AtDCP2が分解活性を担っており,AtDCP1とVCSがその活性を強めることがin vitroの実験により示されている。興味深いことに,この複合体を構成するいずれかの因子を欠失させた変異株は似た表現型を示し,子葉の段階で生育がとまる幼植物致死となる(Xu et al., Plant Cell, 18, 3386-3398, 2006; Iwasaki et al., FEBS Lett., 581, 2455-2459, 2007) 。
5’側からの分解を担う酵素として,シロイヌナズナではAtXRNが報告されている。AtXRN遺伝子は3つの遺伝子ファイミリーから成り,そのうちAtXRN4だけが細胞質に局在することから,この因子が細胞質におけるmRNA代謝に関わる5’-3’ エキソリボヌクレアーゼであると考えられている。atxrn4変異株の解析から,この5’-3’ エキソリボヌクレアーゼが様々なmRNAの代謝に関わっていることがわかっている。例えば,miRNAによって引き起こされるmRNA分解によって生じた,3’側の分解中間産物がAtXRN4により分解されている(Souret et al., Mol. Cell, 15, 173-183, 2004) 。また,RNA依存型RNAポリメレースが関わっているサイレンシングおよび内在遺伝子由来の小分子RNAの合成を負に制御している(Gazzani et al., Science, 306, 1046-1048, 2004; Gregory et al., Dev. Cell., 14, 854-866, 2008) 。植物生理学上の重要な役割として,細菌感染に対する耐性の獲得にも関わっている。Pseudomonas syringaeによって発現が誘導されるlong siRNA (lsiRNA)のひとつであるAtlsiRNA-1は,アンチセンス鎖にある標的遺伝子AtRAPの分解を引き起こす。この分解にAtXRN4が関わっていることが報告されており,AtPARの減少により植物はP. syringaeに対する耐性を獲得することができる(Katiyar-Agarwal et al., Genes Dev., 21, 3123-3134, 2007) 。また,AtXRN4は植物ホルモンのひとつであるエチレンのシグナル伝達経路にも関わっている。エチレンによって発現が誘導される主要な転写因子EIN3は,エチレンからのシグナルが途絶えると,EIN3によって発現が誘導されたEBF1とEBF2の二つのF-boxタンパク質を介したユビキチン/プロテアソーム分解系によって負に制御される。この負のフィードバック制御とAtXRN4によるEBF1/EBF2 mRNAの抑制制御が拮抗することによって,エチレンのシグナル伝達を調節している(Olmedo et al., Proc. Natl. Acad. Sci. USA., 103, 13286-13293, 2006) 。ただし,EBF1/EBF2 mRNAがAtXRN4の直接の標的遺伝子ではないことが示されており,そこにはもっと複雑な制御が存在すると考えられる(Potuschak et al., Plant Cell, 18, 3047-3057, 2006) 。
【3】植物における3’-5’エキソヌクレアーゼ
3’側からの分解はエキソソームという分解複合体が担っている。エキソソームは酵母で詳細な解析が行われており,9個のコアサブユニットから成る環状の構造を持つ複合体に分解活性をもつDis3pが相互作用した形をしている(表1)。核と細胞質の両方に存在し,それぞれが別の相互作用因子と働くことによって,核内の様々なRNAのプロセシングや品質管理,および細胞質では普遍的なmRNA分解に加えて,ある特異的なmRNA分解にも関わっている。シロイヌナズナでは酵母のエキソソームを構成する10個の因子のうちDis3以外の9個の相同因子が複合体を形成していることがわかっている。酵母と違ってシロイヌナズナではAtPPR41が分解活性を持つことから,酵母と植物ではエキソソームの機能構造が異なっていると考えられている(表1)。また,興味深いことに,atrrp41とatrrp4 変異株は致死になるにも関わらず,atcsl4変異株は生育に影響しないなど,構成因子の変異株が異なる表現型を示すことから,酵母のエキソソームとは異なり,それぞれのサブユニットが別個の役割を持つと考えられている(Chekanova et al., Cell, 131, 1340-1353, 2007) 。植物生理学上のエキソソームの役割としては,茎やさやにおけるクチクラワックスの合成の鍵となる遺伝子の発現制御に関わっていること報告されている(Hooker et al., Plant Cell, 19, 904-913, 2007) 。





